Aktuelle Informationen und / oder Hinweise zu den angebotenen Verfahren der Labdia Labordiagnostik GmbH werden hier offengelegt und können mittels angefügten PDF heruntergeladen werden:
Übersicht der angewandten Methoden in der Labdia
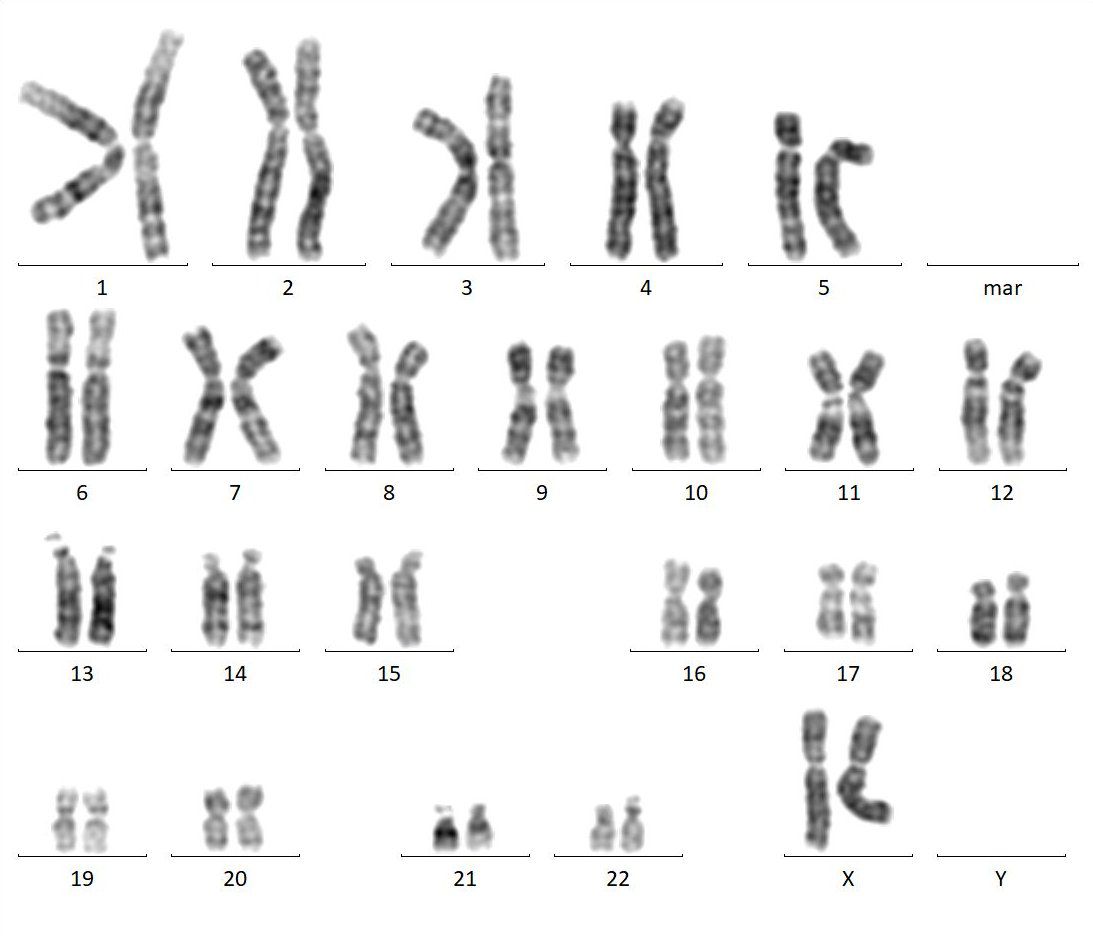
(Molekulare) Zytogenetik
Mit der Chromosomenanalyse oder konventionellen Karyotypisierung können mittels spezieller Bänderungsfärbetechniken die in der Metaphase arretierten Chromosomen sichtbar gemacht und mikroskopisch untersucht werden. So kann mit einer Untersuchung das gesamte menschliche Genom auf numerische und strukturelle Veränderungen überprüft werden, wobei die Auflösung zwischen 5 und 10 Megabasen (Mb) beträgt.
Bei hämatologischen Neoplasien können damit prognostisch relevante somatische Chromosomenaberrationen identifiziert und charakterisiert bzw. auch eine Diagnose gesichert werden. Im Rahmen der Syndromdiagnostik werden mittels der Karyotypisierung angeborene Chromosomenanomalien festgestellt.
Eine international gültige Nomenklatur regelt die Beschreibung normaler, numerisch und strukturell veränderter Karyotypen (ISCN: An International System for Human Cytogenetic Nomenclature).
Auf Anfrage führen wir gerne spezielle Analysen durch und untersuchen andere Tumorentitäten.
Abteilung:
Bei dieser altbewährten Methode werden fluoreszenzmarkierte Gensonden verwendet um chromosomale Veränderungen im Zellkern nachzuweisen.
Dies können Veränderungen der Kopienzahl von einzelnen Genen oder Chromosomen-Abschnitten sein, wie zum Beispiel die Amplifikation des MYCN-Gens oder der 17q Zugewinn bei Neuroblastom sowie 17p (TP53) Verluste bei hämatologischen Neoplasien. Weiters können Translokationen und Fusionen von bestimmten Genen und den damit verbundenen Bruchpunkten detektiert werden (Fusions- und Break apart FISH Sonden), wie zum Beispiel KMT2A (MLL), ABL1, IGH Translokationen.
Mithilfe der FISH kann eine rasche Aussage über das Vorhandensein von bestimmten genetischen Veränderungen getroffen werden, jedoch findet keine Beurteilung des gesamten Genoms statt.
Abteilung:
Mikroskopische Bestimmung der Chromosomenbruchrate anhand PHA (Phytohämagglutinin)-stimulierter Lymphozyten nach Behandlung mit 1,3-Diepoxybutan (DEB), einer DNA-quervernetzenden Substanz („crosslinker“), im Vergleich zu unbehandelten Lymphozyten.
Zellen von Patienten mit Fanconi-Anämie zeigen nach einer DEB-Behandlung eine erhöhte Chromosomenbruchrate und charakteristische Chromatidbrüche bzw. Austauschfiguren aufgrund ihrer erhöhten Chromosomeninstabilität und Hypersensitivität für „DNA-crosslinker“.
Abteilung:
Immunphänotypisierung
Die Durchflusszytometrie bietet die Möglichkeit, verschiedenste Zellen die zuvor mit spezifischen Antikörpern markiert wurden, sicher zu bestimmen, und darüber hinaus sogar zu quantifizieren.
Abteilungen:
Immunzytologie
Bei immunzytologischen Färbungen werden Antikörper zum spezifischen Nachweis von bestimmten Oberflächenmolekülen der Tumorzellen verwendet. Die Detektion erfolgt an einem automatisierten Mikroskop, das Millionen von Zellen in Knochenmarksproben betrachtet und die angefärbten, leuchtenden Tumorzellen darin auffindet. Diese Tumorzellen können dann noch weiter genetisch charakterisiert werden (AIPF = Automated Immunofluorescence Plus FISH). Mit dieser sehr sensitiven Methode kann auch nur eine einzige Tumorzelle unter 1 Million normalen Zellen identifiziert werden, zum Beispiel durch die Verwendung von GD2- und CD56- Antikörpern um Neuroblastom-Zellen nachzuweisen.
Abteilung:
Molekulargenetik
Die PCR (Polymerase Kettenreaktion) ist die Grundlage vieler molekularbiologischer Verfahren. Dabei werden bestimmte DNA Abschnitte im Labor milliardenfach vervielfältigt um diese in weiterer Folge mithilfe verschiedenster Detektionsmethoden sichtbar zu machen. Eine Methode davon ist die Verwendung von Fluoreszenzsonden und die Betrachtung der Vervielfältigung in Echtzeit, wodurch eine genaue Quantifizierung der gebildeten PCR Produkte möglich ist (Realtime-PCR, quantifizierende PCR). Wenn diese Reaktion in tausenden kleinen Öl-Wasser-Tröpfchen abläuft, spricht man von Digital Droplet PCR.
Die Sequenzierung nach Sanger wird verwendet um in bestimmten Gen-Abschnitten jede Base, also jeden Buchstaben des genetischen Codes, zu lesen. Damit können verschiedenste Punktmutationen und auch kleinere Deletionen in diesen Abschnitten nachgewiesen werden. Dies dient dazu die Tumorzellen genauer zu charakterisieren oder die genetische Ursache von Erbkrankheiten zu ermitteln.
Das Next Generation Sequencing (NGS) ist eine neue Technologie, die es ermöglicht viele Sequenzreaktionen parallel ablaufen zu lassen. Damit können einerseits zehntausende Gene des Genoms (Exom) auf einmal analysiert werden, oder auch nur ausgewählte, für die Erkrankung relevante Gene (Panels) sequenziert werden. Diese Methode erlaubt auch die Untersuchung von liquid biopsies (Flüssigbiopsien) bei bestimmten Krebserkrankungen.
Bei SNP Arrays handelt es sich um Mikrochips auf denen Millionen Sonden platziert sind. Damit können hochauflösende Profile der Kopienzahl und Allelverteilung aller Genregionen des menschlichen Genoms erstellt und in weiterer Folge Zugewinne, Verluste und homozygote Abschnitte nachgewiesen werden.
Wir verwenden diese Methode um stratifizierende Marker nachzuweisen, wie zum Beispiel segmentale und numerische Chromosomenaberrationen beim Neuroblastom und den akuten Leukämien. Außerdem wird diese Methode im Rahmen der Syndromdiagnostik eingesetzt.
Die MLPA (Multiplex Ligation dependent Probe Amplification) wird verwendet, um Kopienzahlveränderungen nachzuweisen. Damit können Deletionen oder Duplikationen einzelner oder mehrerer Exons bis hin zu ganzen Genen erfasst werden.
Hämoglobinopathie Analytik
Diese elektrophoretische Auftrennung ermöglicht die Bestimmung von physiologischen und abnormalen Hämoglobinfraktionen im Blut.
Abteilung:
Diese chromatographische Auftrennung des Hämoglobins ermöglicht die Bestimmung von physiologischen und abnormalen Hämoglobinfraktionen im Blut. Sie hat differentialdiagnostische Bedeutung bzw. dient der Hämoglobin-Quantifizierung bei Verlaufskontrollen oder RCE (red cell exchange) z. B. bei bekannter Sichelzellanämie.
Abteilung:
Pharmakologische Analytik
Hochleistungsflüssigkeitchromatographie (engl. high performance liquid chromatography, HPLC) ist ein Verfahren mit der man nicht nur Substanzen trennt, sondern diese auch über Standards identifizieren und quantifizieren (die genaue Konzentration bestimmen) kann.
Abteilung:




